
O químico Guilherme Menegon Arantes: entender como a vida funciona no nível microscópico é um projeto ambicioso da ciência moderna
Nos estudos coordenados por Guilherme Menegon Arantes, o conhecimento de teorias da física, somado ao uso de ferramentas computacionais, ajuda a entender como se dão as reações químicas que coordenam processos biológicos – tudo isso para compreender problemas fundamentais da vida.
“Vida são esses conjuntos de reações acopladas, autorreguladas. Entender o mecanismo dessas reações e seus acoplamentos é entender como a vida funciona no nível microscópico. Este é um projeto ambicioso da ciência moderna”, define Arantes. Professor do Instituto de Química (IQ) da USP, ele coordena o multidisciplinar Laboratório de Bioquímica e Biofísica Computacionais, que apesar de recente na Universidade, vem desenvolvendo uma pesquisa pioneira.
Um dos principais desafios enfrentados pelo grupo é entender como funciona a catálise enzimática. Enzimas são proteínas que controlam a velocidade de reações, como aquelas envolvidas na respiração celular. A presença de uma determinada enzima pode, por exemplo, tornar possível que se dê em apenas um segundo determinada reação química que demoraria um dia inteiro para acontecer. Na ausência de uma enzima, algumas reações seriam tão lentas que seria impossível observá-las no tempo de existência do universo.
Combinando o conhecimento da estrutura molecular da enzima e as teorias de interação das moléculas, os pesquisadores tentam, então, calcular o mecanismo da reação, ou seja, compreender cada etapa que a compõe. Uma reação química pode ser resumida como o reajuste da estrutura dos eletróns de seus reagentes, então o que se busca é saber como acontecem as trocas de elétrons nas enzimas para compreender de que formas elas aceleram as reações.
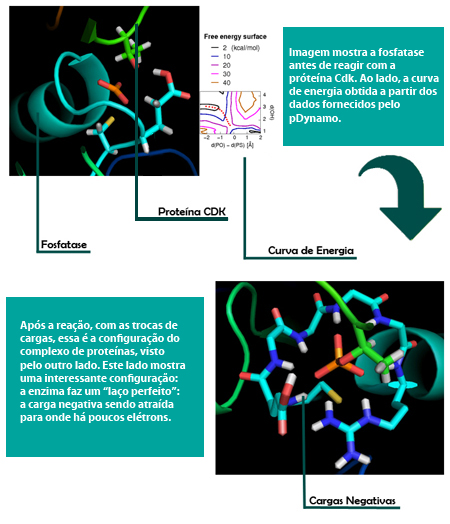
Nesta investigação, os pesquisadores contam com a ajuda de diversas ferramentas computacionais, algumas desenvolvidas com a colaboração do próprio grupo. É o caso do pDynamo, uma biblioteca de programas de código aberto usada para a simulação de sistemas moleculares, em que se pode fazer vários tipos de arranjo de acordo com a estrutura, teoria ou tratamento que se quer dar a uma determinada simulação.
O professor Arantes detalha: “pensamos em um possível modelo de funcionamento da proteína. Então fornecemos a estrutura para o programa, indicando que os átomos estão inicialmente de uma tal forma, e que as interações são dadas por tais equações”. Com essas informações, o programa vai permitir conhecer a energia envolvida nesse processo. A curva de energia obtida pelo pDynamo permite saber qual a velocidade da reação – a partir daí, é possível comparar o resultado dado pelo programa com dados obtidos experimentalmente e saber, então, se o modelo pensado explica a reação.
Diversas outras propriedades também podem ser calculadas na simulação, como a energia necessária para a reação acontecer, ou ainda, a estabilidade dos produtos gerados a partir dela, por exemplo.
O método foi utilizado para conhecer a ação de uma fosfatase, enzima que regula a atividade de outras proteínas com papel importante na divisão celular, ou seja, na produção de novas células. Uma dessas proteínas, chamada Cdk, só permite a passagem para a fase seguinte do ciclo celular na presença da fosfatase.
Plasticidade e flexibilidade: o desafio dos metais
O que atualmente atrai a atenção do professor Guilherme Arantes são as metaloproteínas, ou seja, aquelas que apresentam metais em sua estrutura. Segundo o químico, as teorias e equações para se estudar moléculas orgânicas (compostas, geralmente, apenas por carbono, hidrogênio, nitrogênio e oxigênio) já são bem conhecidas. Já a estrutura eletrônica dos metais é muito mais difícil de ser calculada por sua “plasticidade”, que abre um leque de possibilidades de interação. “Os metais não são bem comportados como o carbono. O ferro, por exemplo, pode facilmente ionizar, perder elétrons externos. O conjunto de possíveis configurações eletrônicas é muito grande e a conta fica difícil”, explica o pesquisador.

Potente, computador do laboratório utiliza ferramentas como o pDynamo para as simulações
Diante dessa dificuldade, Guilherme desenvolveu novas metodologias para o estudo dos metais a partir das observações que vinha fazendo e da leitura sobre pesquisas na área. Com a nova ferramenta teórica, foi possível fazer simulações e entender os experimentos de uma forma que nunca havia sido possível.
O próximo alvo serão as proteínas envolvidas em processos básicos da vida: respiração e fotossíntese. “Processos que envolvem a transformação de um combustível em energia, a respiração celular, e o processo contrário, que é a fotossíntese, são complicados. Quem faz esse meio de campo são as enzimas, principalmente as metaloenzimas. Não é à toa que os metais vem sendo usados pela evolução ao longo do tempo”, afirma o estudioso.
A compreensão sobre a ação enzimática pode dar subsídio, por exemplo, ao desenvolvimento de remédios – conhecendo bem a enzima envolvida em uma determinada infecção, é possível desenvolver um inibidor. O foco do docente, no entanto, é o desenvolvimento de pesquisa básica, estimulado pelo quebra-cabeça teórico que seu objeto de estudo representa. “Meu interesse aqui é um pouco menos aplicado, um pouco menos biomédico, e mais teórico, de curiosidade, de tentar entender o funcionamento de alguns tipos de enzimas porque elas são mais desafiantes”, explica.
Essa curiosidade já rendeu ao pesquisador uma publicação no periódico mais prestigiado da química, o alemão Angewandte Chemie International Edition. Guilherme Arantes organiza agora o pDynamo Workshop & Molecular Simulation, evento que reunirá, no final de novembro, usuários do programa do mundo todo na USP, entre eles seu idealizador, Martin Field, do Institut de Biologie Structurale, na França.
